在人的世界里,孩子的名字往往寄托着父母的希望,也有人喜欢给自己的爱物起一个有寓意的名字。有的科研工作者也会给自己的宝贝研究成果取一个漂亮的名字,这里就不得不提基因编辑界的“劳模”张锋教授。

来源:MIT
张锋教授是CRISPR编辑技术的先驱人物之一,他的名字响当当,成就什么的在这里就不多做赘述了。今天这篇文章中,我们来看看张锋教授给他的科研宝贝们取了哪些漂亮的名字。
1. SHERLOCK
SHERLOCK系统是在2017年4月份首次公布的,论文发表在Science上,这款系统的全称是Specific High Sensitivity Enzymatic Reporter UnLOCKing,直译为“特异性高灵敏度酶解反应器”,是一款以CRISPR-Cas13a为基础开发的检测技术。
常见的CRISPR基因编辑系统使用的是Cas9蛋白,用来切割DNA,而Cas13a则能靶向切割RNA。具体工作原理就是“老司机”向导RNA(gRNA)将Cas13a带到靶RNA前面,Cas13a便会切割靶RNA并释放出预置的荧光信号,也就可以得出检测样本中有某种特定的RNA了,因此SHERLOCK可用来检测RNA病毒感染。
回到名字的话题,根据系统发布的时间我们也可以推测SHERLOCK系统的开发时间与《神探夏洛克》系列剧集和电影的播出时间应该有所重合。

夏洛克·福尔摩斯(来源:《神探夏洛克》剧照)
坦白来说,普通人若是取了刘德华的名字,也会惹人多看几眼。也许是顺了夏洛克的热度,也许他希望所开发的这款系统具有夏洛克一样的敏锐度,又或许他只想表示:我就是个单纯的剧迷or书迷。总之,这个名字,被它妙到了。
再后来,张锋给CRISPR系统的命名就“冷静”了许多,而且更多的是名字与功能的巧妙结合。
2. REPAIR
第二个系统是2017年10月发布于Science的REPAIR系统,名字来源于RNA Editing for Programmable A to I Replacement,直译为“可编程的A到I替换的RNA编辑”。
如果说SHERLOCK是一把RNA砍刀,那么REPAIR就是一个定点修补刀,因为REPAIR可以编辑RNA,完成RNA编辑用的是内源性RNA腺苷脱氨酶(ADAR),这是一种天然酶,能将RNA上的腺苷(A)转变为肌苷(I)。
在REPAIR系统中,ADAR不是单独行动的,它与Cas13b蛋白进行了融合,Cas13b蛋白与之前提到的Cas9和Cas13a都属于Cas蛋白家族。与ADAR融合的Cas13b经过了一些特殊改动,失去了剪切能力,但保留了靶向RNA的活性。
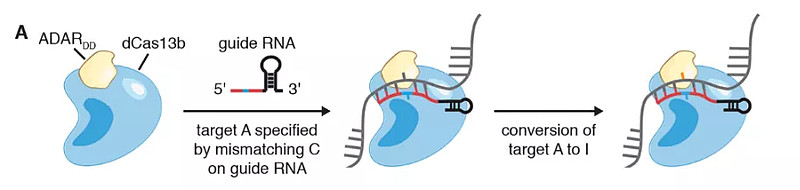
REPAIR系统构造与功能(来源:参考资料[2])
在编辑过程中,REPAIR可以将特定位点上突变的腺苷(A)纠正为肌苷(I),而肌苷会被翻译器“读”为鸟苷(G),因此,REPAIR很有希望治疗一些发生G→A突变的疾病。
如果说某些疾病的发生是因为身体的某些零部件发生了错误,那么REPAIR系统的目标就是修复这些错误,使机体恢复正常的运转,这一功能也与repair这个词相得益彰。
3. RESCUE
RESCUE系统是在2019年7月份公布的,研究发表在Science上,也是一款RNA编辑器,全称RNA Editing for Specific C to U Exchange,即“特定C到U交换的RNA编辑”,从中也可看出这款编辑器的功能是完成胞苷(C)到尿苷(U)的转变。
RESCUE是REPAIR的升级版,升级方法是酶进化,机制略复杂这里就不多赘述了,关键点在于升级后的RESCUE增加了一个新的编辑方向:将C转变为U,也就是说RESCUE能进行A→I和C→U的多重编辑。
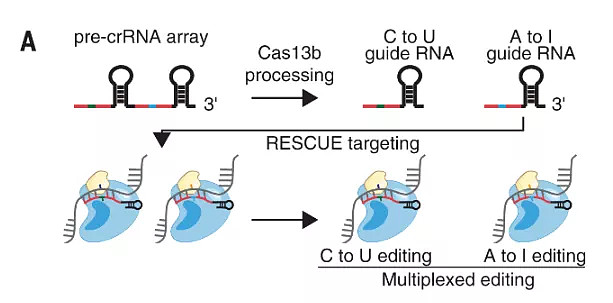
RESCUE系统构造与功能(来源:参考资料[3])
以APOE4基因的编辑为例讲一下RESCUE的功能。APOE4是一种阿尔茨海默症相关的风险基因,它还有一个“胞兄”APOE2,APOE2与APOE4只有两个碱基的区别,APOE4 RNA中两个C对应APOE2的两个U,两者编码的蛋白相差无几,但是APOE2是无害的。利用RESCUE系统能把APOE4 RNA分子中的C转为U,将危险的APOE4转为无害的APOE2。
所以RESCUE的作用就好比拆除定时炸弹,排除风险,这和REPAIR的取名方法十分相似。
4. STOP
再来看第4款STOP,你可以先猜一猜它的含义。根据上面介绍的REPAIR和RESCUE的命名规律,STOP应该是一种用于阻止感染或者阻止疾病进展的系统。
答案是前者,STOP是用来检测新冠病毒感染的系统,名字来源于Sherlock Testing in One Pot,也就是“Sherlock在一个容器里测试”,是改良版的SHERLOCK系统,公布于2020年5月份。因为SHERLOCK检测病毒的时候有多个处理步骤,多次样本转移可能会造成污染,而将SHERLOCK的化学反应步骤集中在同一个试管中进行,就能大幅减少这种污染。
STOP使用了环介导等温扩增技术(Loop-mediated Isothermal Amplification, LAMP)扩增RNA,为了匹配这一技术60℃的反应温度,Cas13a酶被替换为AapCas12b酶,再加上对gRNA和添加剂等的优化和RNA提取的简化,STOP检测新冠病毒样本时表现出了高特异性(100%)和高灵敏度(97%),而且STOP可以在1小时内得到检测结果。
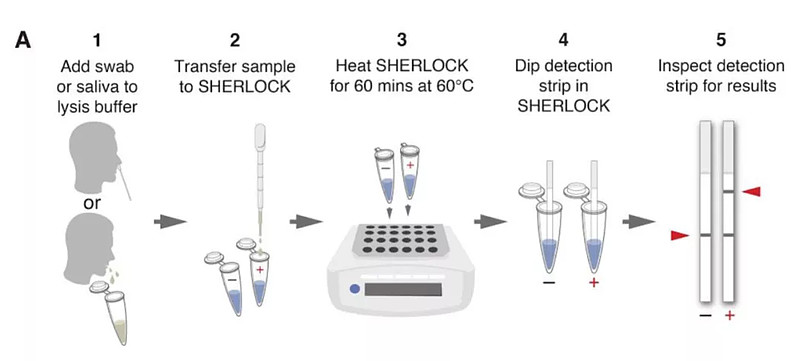
STOP的检测步骤(来源:参考资料[4])
STOP虽然名字与功能看起来没有像REPAIR和RESCUE与功能那么的契合,但也符合研究者想要通过提高新冠病毒感染检测的速度和精确性来为抗疫做贡献,希望阻止病毒的传播。
5. SEND
SEND就不是CRISPR系统了,但是开发初衷是为了递送CRISPR,相关研究今年8月份发表在Science上。SEND全称Selective Endogenous eNcapsidation for cellular Delivery,即“选择性内源性包裹用于细胞递送”。
在这里我们先讲一下CRISPR系统的递送,常用的一种递送方式是将系统的原件以RNA的形式递送到胞内,在体内再翻译和组装成一个完整的编辑系统,一个优秀的递送系统要能把CRISPR系统的零部件RNA有效递送到特定位点。
SEND系统的开发灵感来源于一类古老的基因,它们在远古时期并不属于人类,而是属于某种病毒,这些病毒在感染了人体后把自身的基因组整合到了人的基因组中,在数百万年的进化中逐渐被人体吸收,成为了人体的一份子。
PEG10就是其中一种,它还有一个特性,PEG10蛋白能与编码PEG10蛋白的mRNA结合并促进PEG10 mRNA以囊泡的形式分泌。也就是说,这个蛋白能自动包装和转移mRNA。
有了这一基础,研究人员先确定了能触发这一过程的最小序列,然后利用这一序列设计了PEG10和想要递送的RNA,以便PEG10可以选择性地包装这些RNA。然后再对形成的囊泡修饰修饰,增加囊泡的靶向特异性。这样就能把RNA递送到特定的细胞、组织或器官中了。
在实验中,SEND也成功地将CRISPR系统的Cas9和sgRNA递送到了需要进行编辑的地方,SEND这个名字也同样与系统的功能十分相称。
6. SLEEK
最后一款SLEEK是Editas Medicine在今年8月份公布的,Editas是张锋教授创办的公司。SLEEK系统主打能对诱导多能干细胞(iPSC)进行100%的基因敲入(knock-in),全称SeLection by Essential-gene Exon Knock-in,即“必需基因外显子敲入筛选”。
乍一看这个描述似乎没有上面几款系统那么直白,“筛选”是啥?“必须基因外显子”又是啥?我们先来看“筛选”是什么含义。
在现在热门的CAR-T、CAR-NK细胞疗法的开发中,很多都利用了CRISPR基因编辑工具进行基因的改造。但CRISPR在改造细胞基因的过程中,有可能没有敲入转基因,而是引入一种称为“indel”的基因突变。
SLEEK就是针对indel基因突变开发的,经SLEEK编辑改造后的细胞,能自动筛选敲入的转基因,如果是需要的转基因,细胞活,如果是indel,细胞则死,这样筛选过来剩下的细胞就基本都是成功敲入转基因的细胞了。
再来看“必需基因外显子”的作用。上面讲插入indel基因突变细胞会死,那是因为SLEEK把基因编辑位点选在了某个重要(必需)基因的外显子上,indel插入后这个必须基因无法表达,细胞因此死亡,但SLEEK巧妙的一点是它引入的基因片段能维持这个必需基因正常表达的序列,因此如果转基因引入成功,必需基因和转基因都能正常表达,这样SLEEK就可以完成筛选和排除突变的作用。
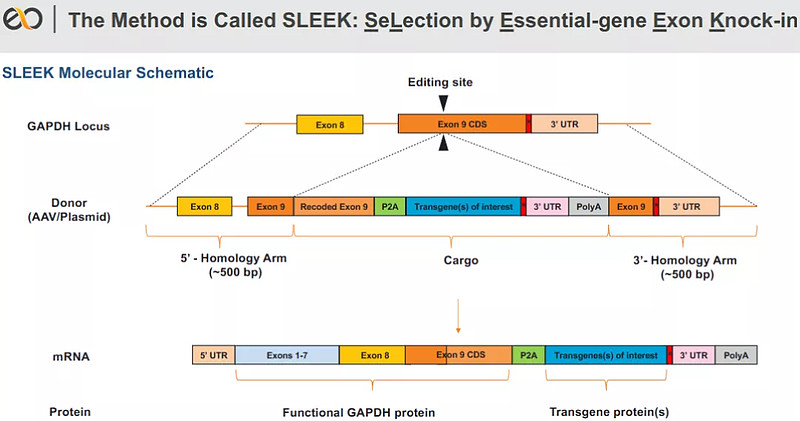
SLEEK的编辑机制,GAPDH是糖酵解反应的一个关键酶(来源:参考资料[6])
现在这一方法已成功在iPSC细胞、T细胞和NK细胞中进行了测试,效率嘛当然没有达到100%,而是接近100%,100%敲入应该是这一系统优化的目标。
解释了机理,再来看一下功能与名字的适配度,sleek的形容词含义是“光滑的、线条流畅的”,动词含义是“(使)平整、光亮”。这么看,难道SLEEK这个系统的大名应该是研究者对敲入基因的细胞的希望:光滑,不突变?这是中文语境的理解,不太严谨,换做英文语境,有可能是研究者希望系统的敲入顺滑。
讲到这里,张锋教授的6款系统就介绍完了,花样繁多,名字与功能齐飞,那么哪一款或哪几款系统的名字最深得你心呢?
还有哪些科学家们喜欢给自己心爱的科研成果起一个漂亮的名字呢?欢迎大家留言。
参考资料:
[1] Gootenberg J.S., Abudayyeh O.O., Lee J.W., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science (2017)
[2] Cox D., Gootenberg J.S., AbudayyehO.O., et al. RNA editing with CRISPR-Cas13. Science (2017)
[3] Abudayyeh O.O., Gootenberg J.S.,Franklin B., et al. A cytosine deaminase for programmable single-base RNAediting. Science (2019)
[4]网页链接
[5] Segel M., Lash B., Song J.W., et al.Mammalian retrovirus-like protein PEG10 packages its own mRNA and can bepseudotyped for mRNA delivery. Science (2021)
[6] 网页链接