
$Illumina(ILMN)$ $太平洋生物科学(PACB)$ $ont.l$ $Singular Genomics(OMIC)$
Illumina宣布推出Infinity进入长读长测序领域,虽然在具体技术细节上缄口不言,但最近在不同场合也展示了Infinity的案例。
在这个十几分钟的主题报告中,来自 Illumina的Gary Schroth 博士通过基因浏览器IGV屏幕截图的形式展示了Infinity长读长与市面上其他长读长技术的比较。这里的市面上长读长技术无疑指的就是Pacbio的循环共识测序(CCS)。(美国市场对于这两家公司来说都是主导,所以针锋相对也很正常;而与牛津纳米孔技术相比在读长也没有太大优势,所以直接略过了ONT。)

展示武艺,同台竞技,自然被对手关注到了。Pacbio公司随后就写了自己的观后感:首先感谢你帮我们又证明了HiFi 测序优越的数据质量,但不得不说Infinity 合成长读取有重大错误并引入了不少人为问题。
偷鸡不成蚀把米?
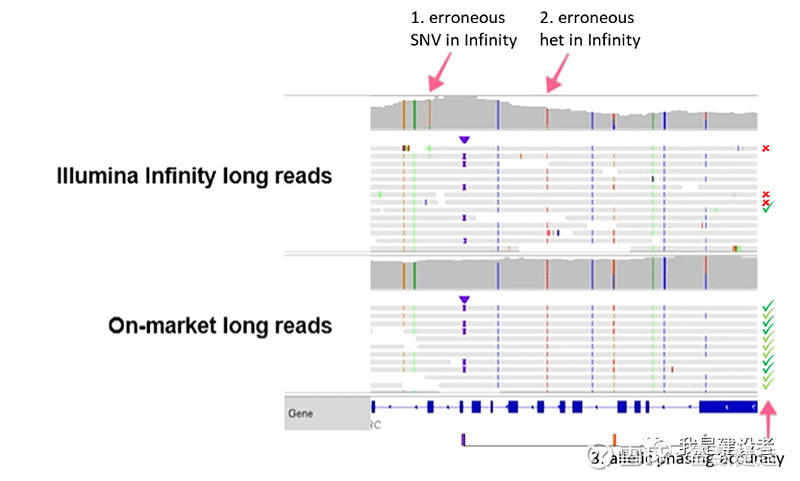
Pacbio指出,如STRC基因这个例子提示,Infinity 数据中有几处错误(如下图中所示):包括(1)假阳性 SNV、(2)被错误地称为杂合子的纯合子 SNV、(3)三个杂合变异的不正确定相(由于 Infinity 读取长度不足以跨越该区域,因此10/14个读取中没有提供信息,而剩下的4个读取行中三个给出了错误答案。)
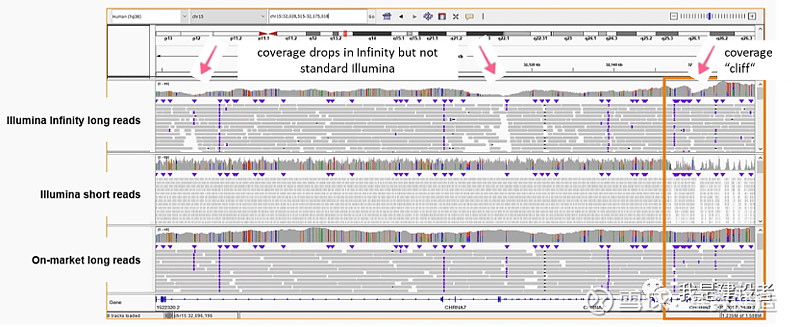
而如另一个CHRNA7基因的例子提示,Infinity 数据中看到覆盖率的两次显着下降,提示这些覆盖缺口应该是通过合成长读取引入的(因为这两个区域被标准的 Illumina 测序所覆盖)。可以在 Infinity 数据中也看得到典型的覆盖“悬崖”,提示了数据质量的问题。
Pacbio在之前的回应中也指出过类似的问题,包括由于Infinity 仅部分检测到,使得变异错误地显示为杂合子;由于这样的读取不足,可能造成变异识别的置信度问题;在长距定相上,由于读长的限制,不能直接定相。

以上这些问题,的确是比较清晰可见,反应了Infinity技术上的局限性,这些一定也是Illumina在持续优化解决的方面。
随着Illumina 宣布推出Infinity,Element Biosciences 收购了 Loop Genomics,Singular Genomics宣布推出XR-seq,在传统短读测序平台上实现合成长读长变成了一个明确开发选项。上文说:Illumina 进入长读长标志了 DNA 测序市场的转变。没错,这个趋势日渐明显:追求长读长,或者说实现10kb读长即将变成进入短读测序平台竞争的门槛。
与天然长读相比,即便合成长读长暴露这些弊端,合成长读长技术即便只是增加到10kb的读取,也可以帮助解决一定的科学或临床诊断问题。同时,考虑到成本与收益,可及可负担的合成长读长不失为一个有效手段。但随着Pacbio和ONT不断提升通量、降低测序成本、并不断提升测序的准确率(包括不同读长),这样的技术成本vs潜在收益的衡量一定也会逐渐调整。