2021年6月2日,David Liu 实验室又在Nature杂志上发表了一篇单碱基编辑器治疗小鼠SCD的论文。
之前我的文章有讲到过BEAM在研发BEAM-102时PAM还是有很大的限制。那时PAM主要有NGG,NGC和NGA三种。而编辑SCD Makassar只有用NGC PAM比较合适,编辑位置为9,最初的经典ABE是无法高效的编辑这个位置的A的。BEAM改用IBE后,这个问题得到了一定的解决,配合ABE8,编辑效率可以达到70~80%。但其实最理想的方案其实还是能将目标A置于位置7,这样编辑效率会达到最高,不过要实现这个来编辑SCD需要靶向CACC的PAM,当时的BEAM并没有这个选项。不过后来David Liu实验室采用定向进化的方法,于2020年发表了新型的PAM,可以扩展到NR,R为G或者A。今天David Liu实验室在Nature上发表的这篇论文里面用的就是 ABE8e + NRCH PAM(R为A或G,H为A或C或T)。
但是看完了这篇论文,我的心情其实是喜忧参半。先来说说好的部分,那就是ABE8e + NRCH PAM(目标的PAM序列为CACC)确实能将需要编辑的目标A移到位置7,能在人类HPSC细胞里做到80%的编辑效率,同时几乎没有missense的bystander。

但是将人类SCD患者的HSPC细胞移植到小鼠后,编辑效率从80%下降到了68%(这是个潜在的小问题),移植后从小鼠体内提取的细胞显示HbS从96%下降到了40%。
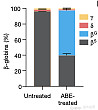
这个结果和BEAM-102体外细胞试验中HbS从100%下降到了10%是有明显差距的。我们目前也还不清楚当BEAM-102移植到体内后,HbS能否还保持在10%这个低水平。
由于人类的红细胞在小鼠体内生存时间比较短,David Liu实验室还做了另外一个试验,那就是将小鼠SCD模型的HSPC编辑过后移植到SCD小鼠体内。在这个试验中,移植前的编辑效率为53%,移植后降低到44%,但是在小鼠体内循环流通的红细胞中,HbG的比例却高达79%。这很有可能是因为含有HbG的红细胞的平均寿命要远高于含有HbS的红细胞。后续进一步的试验发现,只要HbG的编辑效率到达了20%以上,循环流通的红细胞中HbG的比例即可达到80%左右,血液中的其它各项指标也接近于健康的小鼠。
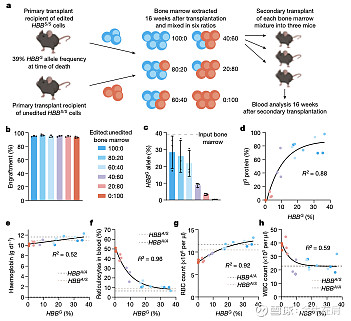
尽管以上的结果还是相当不错的,但也出现了一个重大隐患:应用NRCH的PAM后虽然可以编辑以前“够不着”的目标,但是靶向的特异性也因此大打折扣。在接下来的脱靶效应分析中发现了大量的guide dependent的脱靶现象。具体来说在725个潜在的脱靶位置中(有697个可以测序)其中有7.8%的位置(54/697)出现了脱靶现象(对比在不久前Verve的PCSK9人类肝脏细胞试验中,只有69个潜在的脱靶位置,而且最后无一出现真正可观察到的脱靶现象)。虽然后续分析显示这54个脱靶位置大都不在重要位置,应该不太可能造成临床后果,但是NRCH PAM如此严重的脱靶恐怕还是无法直接进入临床的。后续必须进一步进化形成专门靶向CACC的PAM才让人更放心。
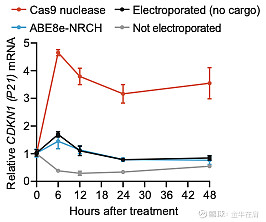
这篇文章后面还有一个关于用Cas9-nuclease和ABE进行基因编辑安全方面的对比也非常有趣。文中以CRSP的SCD项目中编辑(破坏)BCL11A基因为例,将其造成的后果来和ABE的对比。首先是对比了p53-mediated DNA damage response(通过测量CDKN1基因的表达)。一旦某种原因造成了DSB,正常细胞一般会停止分裂,p53启动,等修复完DSB后再继续分裂。但是如果某个细胞的p53基因有缺陷的话,细胞有可能会无视DNA的损伤继续分裂,造成细胞染色体组的进一步损伤,这会极大的增加致癌的风险。因此大规模p53反应是会有利于潜在癌变细胞的增长的,增加其相对于正常细胞的比例。由下图可以看到,Cas9-nuclease会造成p53反应,而ABE的编辑本身则并不会造成p53反应(Electroporation递送是会有一定的p53反应)。
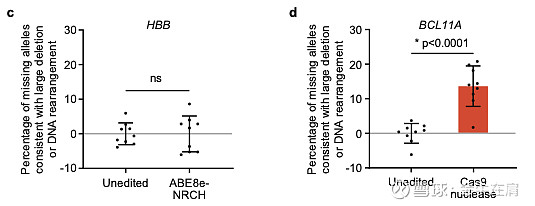
Cas9-nuclease 编辑另一个更加惊人的风险是会造成基因大量的 long deletions or rearrangements。比如编辑BCL11A基因时,就有高达14%的 long deletions or rearrangements,而用ABE编辑时则没有发现这个问题。这个比例有点恐怖了,如果编辑的基因附近正好有其它重要的基因,后果可能难以想象。
最后总结一下:
一,NRCH及类似的PAM的特异性和更传统的PAM(如NGG,NGC,NGA)比还不够好,因此PAM还是有可能限制单碱基编辑器的编辑目标范围的。BEAM今后看来还是得针对具体的目标来进化出专门的PAM才行。
二,之前BEAM-101和Verve-101报告的没有脱靶现象看来并没有那么容易做到。我们不能简单的认为今后的项目也一定没有guide dependent的脱靶效应。
三,Cas9-nuclease编辑看来比想象中的风险要高很多,居然发生了这么高比例的 long deletions or rearrangements,看来那些依赖于Cas9-nuclease编辑的公司之前都没有透露出其真实的风险。
另外,对于要依赖于递送DNA的疗法,比如 gene therapy 或者是 Cas9+HDR 或者是 Cas9+NHEJ mediated DNA insertion都是有潜在风险的。因为凡是递送DNA进入细胞内都是有一定的概率会发生 insertional mutagenesis 的。