今天 (2021年10月14日),David Liu 实验室发表了其新一代 Prime Editor 的具体改进细节的后半部分,被命名为 PE4max 和 PE5max。
他们首先用 Repair-seq CRISPRi 系统性的检查了可能影响 Prime Editing 系统的 476 种基因。他们发现其中 DNA 错配修复(MMR)通路会大幅的抑制 Prime Editing 系统的编辑效率并且会增加 Indel 和其它副产品的产生。他们发现 MMR 会发现 Prime Editing 过程的中间产物 — 异源双链体,剪切掉因为编辑而产生的断裂了的单链,从而使编辑失效。
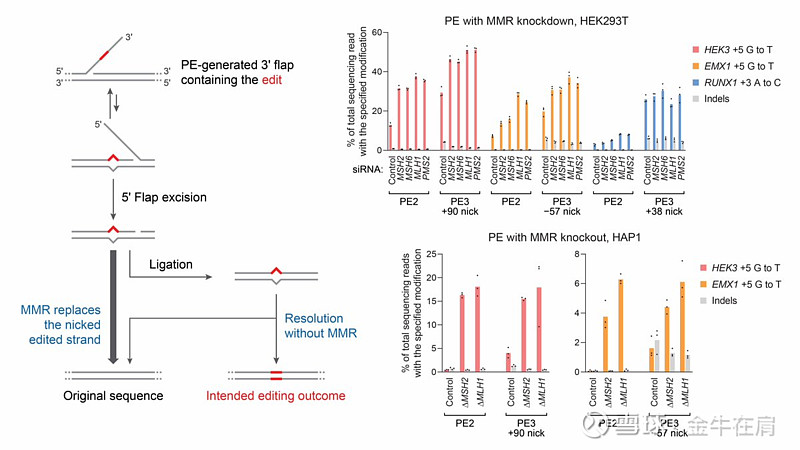
他们设想能不能通过敲除或者降低 MMR 相关基因的表达来提高 PE 的编辑效率呢。于是他们尝试了三种不同的办法,第一种是瞬时表达 MLH1 dominant negative (MLH1dn) 基因,第二种是表达 MLH N端的蛋白来抑制其 MSH2 binding,第三种则是将 MLH1dn 蛋白和 PE2 融合在一起形成 PE2-P2A-MLH1dn。最后结果发现用第一种方法的效果最好。尽管如此,敲除 MLH1 蛋白的话效果还会更好,因此未来可能还会找到更好的办法来抑制 MMR 通路。
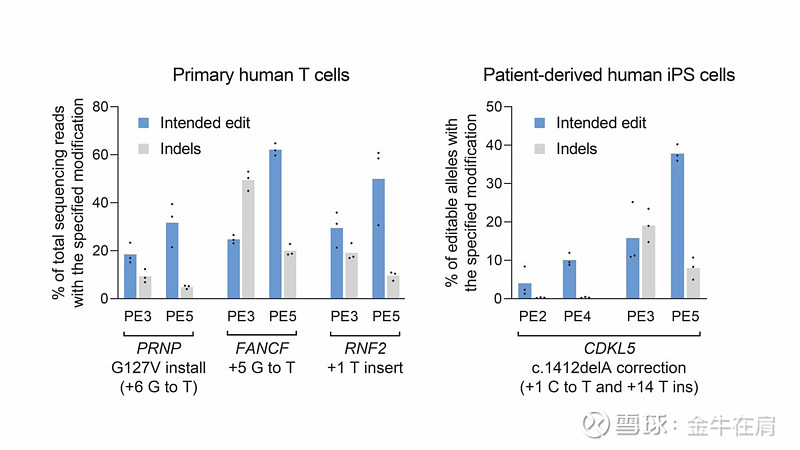
于是我们有了新一代的 Prime Editor PE4 (PE2+MLH1dn) 和 PE5 (PE3+MLH1dn),其不仅编辑效率能有大幅增加,其 Indel 的产生率也都有所降低。现在 PE5 可以在 Primary T Cells 中做到 30~60% 的编辑效率。需要注意的是 PE5 由于需要切断对面的单链,因此还是会有不少的 Indel 产生。与此对应的是 PE4,由于其不切断对面的单链,Indel 率一般不超过1%,但编辑效率会有不小的牺牲。
有趣的是,之前发现 PE2/PE3 在 HEK293T 细胞中的编辑效率普遍高于在 HeLa,K562 以及 U2OS 细胞中的编辑效率是因为 HEK293T 细胞中的 MLH1基因的启动子已被高度甲基化而抑制了其表达,而在其它几种细胞中 MLH1 则是正常表达。
由于MMR是非常重要的修复DNA的通路,尤其是可以抑制 microsatellite instability (MMR先天缺失单份的话会极大的增加大肠癌以及其它很多癌症的发病率),以及修复被误复制而导致的单碱基突变(概率降低一百倍左右),因此瞬时表达 MLH1 dominant negative (MLH1dn) 基因是有一定的风险的。但好在研究发现如果只是瞬时表达几天的话并不会检测出对 microsatellite instability 的影响。

研究人员还发现,MMR 通路并不是对所有的单碱基突变都非常敏感,其不太能发现 G 到 C 的单碱基突变,因此用原来的 PE2/PE3 来实现该编辑的效率会普遍较高。利用这一点,研究人员可以通过有选择性的引入一些 silent or benign mutation 到目标编辑位置附近产生一个MMR相对不敏感的突变,这样即使用原来的 PE2/PE3 也能实现较高的编辑效率。
最后,他们还进一步的优化了 Cas9 蛋白,入核信号,链接构成,以及密码子,再加上之前优化过的 epegRNA 来进一步的提高 PE4/PE5 系统的编辑效率,命名为 PE4max/PE5max。
总的来说,这次 PE 系统的改进还是非常值得称道的,尤其是明确了影响之前编辑效率的重要的DNA修复的通路,这也为以后的进一步优化也打下了基础。另外,MMR 通路一般只影响较短序列的 mismatch,超过13 bp 后几乎完全不起作用,这也暗示了可能还有其它DNA修复的通路会对长序列的删除和插入产生抑制作用。我们不久以后应该还会听到关于 PE 改进的新消息。